L’année dernière, à Ignite, Microsoft a annoncé une offre cloud native avec Azure Container Apps, permettant aux développeurs de créer des architectures de microservices à l’aide de conteneurs en avant-première en mode Serverless. La société a maintenant annoncé la disponibilité générale du service lors de la conférence annuelle Build.
Azure Container Apps repose sur une technologie open source avec des projets CNCF tels que Kubernetes Event-Driven Autoscaling (KEDA) , Distributed Application Runtime (Dapr) et Envoy s’exécutant sur Azure Kubernetes Service (AKS).
Conteneurs dans Azure Container Apps
La version générale d’Azure Container Apps inclut désormais de nouvelles fonctionnalités telles que la possibilité pour les développeurs de sécuriser leurs applications en les déployant sur un réseau virtuel à l’aide d’ identités managées pour accéder à d’autres ressources protégées Azure Active Directory (Azure AD).
De plus, ils peuvent configurer l’authentification et l’autorisation intégrées pour leurs applications externes. De plus, des domaines et des certificats personnalisés peuvent être utilisés pour personnaliser les applications. En outre, diffusion en temps réel des journaux pour les messages de journal stdout et stderr, connexion aux consoles du conteneur , examen des métriques et définition des alertes peuvent tous être utilisés pour surveiller la santé et les performances des applications.
Azure Container Apps peut être utilisé dans divers contextes, des points de terminaison d’API publics aux microservices. Le service permet l’exécution de code d’application encapsulé dans n’importe quel conteneur, quel que soit l’environnement d’exécution ou le modèle de programmation. De plus, le code peut être développé dans par exemple avec Visual Studio Code, le portail Azure et Azure CLI pour gérer les ressources Azure Container Apps.
Je vais ici faire un test via le portail Azure car la partie CLI est très bien documentée ici :
Quickstart: Deploy your first container app
puisque Azure Container Apps est disponible depuis le marketplace :
Applications Azure Container | Microsoft Azure
Je définis le nom du Resource Group ainsi que celui du conteneur depuis le portail :
Les valeurs par défaut de l’environnement notamment pour la partie réseau et monitoring sont laissées ici en l’état :
Je réutilise une image officielle docker de JupyterLab dans sa version Datascience (qui comprend Python, Julia et R) via les caractéristiques du conteneur à renseigner dans le portail :
Avec ici 2 vCPU et 4 Go de mémoire vive. Voir à ce propos les caractéristiques de la tarification d’Azure Container Apps avec 180,000 vCPU-secondes et 360,000 GiB-secondes gratuites par mois :
Azure Container Apps - Pricing | Microsoft Azure
Azure Container Apps expose chaque application par le biais d’un nom de domaine si Ingress est activé. Les points de terminaison Ingress peuvent être exposés publiquement au monde entier ou en interne et uniquement disponibles pour les autres applications de conteneur dans le même environnement. J’active cette option dans le portail avec le port TCP 8888 comme port cible pour le conteneur JupyterLab :
Connect applications in Azure Container Apps
L’application avec JupyterLab est prête à être lancée :
Déploiement effectué avec succès ici :
Par défaut, je dispose d’un min/max dans le Pod dans AKS avec 0 à 10 réplicas du conteneur :
Je passe à un seul conteneur ici :
Ce qui entraîne la création d’une révision :
avec cette nouvelle valeur :
Comme il y a un seul conteneur en exécution ici, je récupère le token nécessaire à la connexion à JupyterLab dans la partie Log Stream du portail :
En effet, l’ingress a été fournie dans le portail :
toujours sous cette forme :
Connexion à JupyterLab :
Je dispose ici de la dernière version de Julia 1.7.3, ce qui me sera utile pour tester les modèles de substitution via Surrogates.jl.
Comme l’indique ce dépôt sur GitHub, un modèle de substitution est une méthode d’approximation qui imite le comportement d’une simulation coûteuse en termes de calcul. En termes plus mathématiques : supposons que nous essayons d’optimiser une fonction f(p), mais que chaque calcul de f est très coûteux. Il se peut que nous devions résoudre une EDP (équation aux dérivées partielles) pour chaque point ou utiliser des machines d’algèbre linéaire numérique avancées, ce qui est généralement coûteux. L’idée est alors de développer un modèle de substitution g qui se rapproche de f en s’entraînant sur des données précédentes collectées à partir d’évaluations de f. La construction d’un modèle de substitution peut être considérée comme un processus en trois étapes :
- Sélection de l’échantillon
- Construction du modèle de substitution
- Optimisation du modèle de substitution

https://www.researchgate.net/publication/338658899_Surrogate-assisted_global_sensitivity_analysis_an_overview
Je peux créer un nouveau kernel prenant en compte le nombre de threads présents, ici au nombre de 4 :
using IJulia
installkernel("Julia-4-threads", env=Dict("JULIA_NUM_THREADS"=>"4"))
Un nouveau Kernel avec les 4 threads pour Julia est alors disponible :
J’utilise ici bottom en lieu et place d’htop pour surveiller la consommation de ressources dans JupyterLab :
GitHub - ClementTsang/bottom: Yet another cross-platform graphical process/system monitor.
avec le thème Jupyter Dark pour une meilleure visualisation dans JupyterLab:
Installation du paquet Surrogates.jl et Plots.jl ensuite :
using Pkg
Pkg.add(["Surrogates", "Plots"])
Application ici à la fonction de Branin qui est couramment utilisée comme fonction de test pour la métamodélisation dans les expériences informatiques, notamment dans le contexte de l’optimisation :
avec
J’importe les paquets ici :
using Surrogates
using Plots
default()
et je définis la fonction :
function branin(x)
x1 = x[1]
x2 = x[2]
b = 5.1 / (4*pi^2);
c = 5/pi;
r = 6;
a = 1;
s = 10;
t = 1 / (8*pi);
term1 = a * (x2 - b*x1^2 + c*x1 - r)^2;
term2 = s*(1-t)*cos(x1);
y = term1 + term2 + s;
end
avec la réprésentation graphique de cette fonction :
n_samples = 80
lower_bound = [-5, 0]
upper_bound = [10,15]
xys = sample(n_samples, lower_bound, upper_bound, SobolSample())
zs = branin.(xys);
x, y = -5:10, 0:15 # hide
p1 = surface(x, y, (x1,x2) -> branin((x1,x2)))
xs = [xy[1] for xy in xys]
ys = [xy[2] for xy in xys]
scatter!(xs, ys, zs)
p2 = contour(x, y, (x1,x2) -> branin((x1,x2)))
scatter!(xs, ys)
plot(p1, p2, title="Fonction vraie")
ce qui donne ce graphe :
Il est maintenant temps d’ajuster différents substituts et de les tracer. Application à la substitution par krigeage.
Le krigeage ou régression par processus gaussien est une méthode d’interpolation pour laquelle les valeurs interpolées sont modélisées par un processus gaussien.
kriging_surrogate = Kriging(xys, zs, lower_bound, upper_bound, p=[1.9, 1.9])
et de sa représentation graphique :
p1 = surface(x, y, (x, y) -> kriging_surrogate([x y]))
scatter!(xs, ys, zs, marker_z=zs)
p2 = contour(x, y, (x, y) -> kriging_surrogate([x y]))
scatter!(xs, ys, marker_z=zs)
plot(p1, p2, title="Substitution par Krigreage")
Utilisation ensuite du “modèle de substitution à distance inverse”.
Il s’agit d’une méthode d’interpolation. Dans cette méthode, les points inconnus sont calculés avec une moyenne pondérée des points d’échantillonnage.
Ce modèle utilise la distance inverse entre les points inconnus et les points d’apprentissage pour prédire le point inconnu. Nous n’avons pas besoin d’ajuster ce modèle car la réponse d’un point inconnu x est calculée par rapport à la distance entre x et les points d’apprentissage.
InverseDistance · Surrogates.jl
InverseDistance = InverseDistanceSurrogate(xys, zs, lower_bound, upper_bound)
et encore une fois sa représentation graphique :
p1 = surface(x, y, (x, y) -> InverseDistance([x y]))
scatter!(xs, ys, zs, marker_z=zs)
p2 = contour(x, y, (x, y) -> InverseDistance([x y]))
scatter!(xs, ys, marker_z=zs)
plot(p1, p2, title="substitution distance inverse")
Utilisation enfin de la substitution de Lobachevsky. La fonction splines Lobachevsky est une fonction utilisée pour l’interpolation dispersée univariée et multivariée. Introduit par Lobachevsky en 1842 pour étudier les erreurs dans les mesures astronomiques :
Lobachevsky = LobachevskySurrogate(xys, zs, lower_bound, upper_bound, alpha = [2.8,2.8], n=8)
accompagnée de sa représentation graphique :
p1 = surface(x, y, (x, y) -> Lobachevsky([x y]))
scatter!(xs, ys, zs, marker_z=zs)
p2 = contour(x, y, (x, y) -> Lobachevsky([x y]))
scatter!(xs, ys, marker_z=zs)
plot(p1, p2, title="Substitution Lobachevsky")
Il est possible de définir un réseau de neurones comme modèle de substitution, en utilisant Flux.jl :
En effet, il est possible d’appeler des méthodes d’optimisation via les réseaux neuronaux. Surrogates.jl se retrouve dans JuliaSim, une plateforme de simulation de nouvelle génération basée sur le cloud, combinant les dernières techniques de SciML avec la modélisation et la simulation de jumeaux numériques basés sur des équations :
comme le décrit cet article …
Pour finir, je peux reprendre l’utilisation de ParallelStencil.jl comme illustré dans l’article précédent :
- Parallélisation distribuée presque triviale d’applications GPU et CPU basées sur des Stencils avec…
- GitHub - omlins/ParallelStencil.jl: Package for writing high-level code for parallel high-performance stencil computations that can be deployed on both GPUs and CPUs
en installant les paquets nécessaires :
Pkg.add(["Printf", "Statistics", "ParallelStencil"])
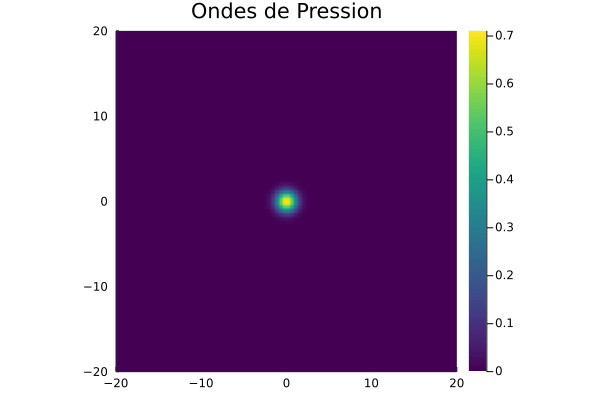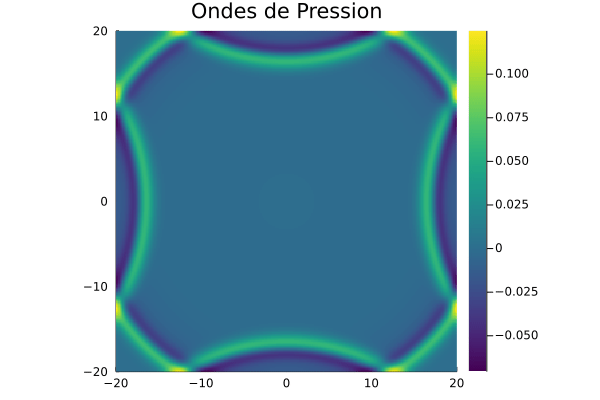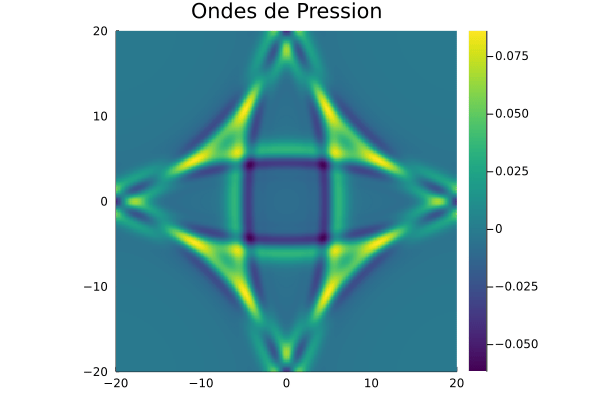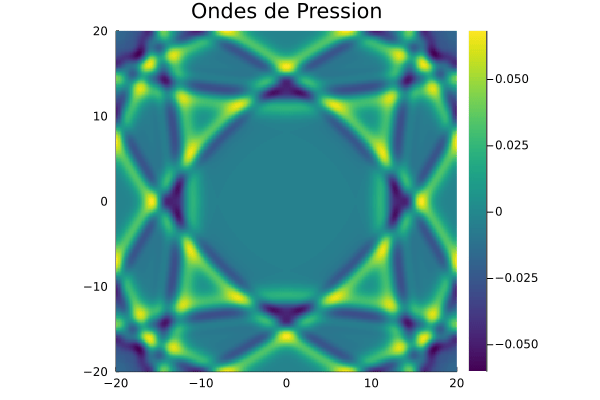
avec l’exemple des ondes acoustiques en 2-D en utilisant la formulation de vitesse-pression fractionnée :

https://pt.slideshare.net/AmrMousa12/2-dimensional-wave-equation-analytical-and-numerical-solution
et ce code en Julia :
Avec un suivi de métriques de consommation des ressources depuis le portail :
ou depuis le conteneur via bottom :
La suppression de JupyterLab se fait simplement par la suppression du Ressource Group crée dans le cadre d’Azure Container Apps, via le portail ou Azure CLI :
az group delete --resource-group RG-JUPYTER --yes
Pour conclure, on peut aller beaucoup plus loin dans Azure Container Apps avec Dapr (Distributed Application Runtime), un runtime qui permet de créer des microservices résilients, sans état et avec état, qui peut être mis en oeuvre dans ce cas …
Tutorial: Deploy a Dapr application to Azure Container Apps using the Azure CLI
Avec Azure Container Apps, vous obtenez une version entièrement gérée des API Dapr lors de la création de microservices.
Lorsque vous utilisez Dapr dans Azure Container Apps, vous pouvez permettre aux side-cars de s’exécuter à côté de vos microservices qui fournissent un riche ensemble de fonctionnalités.
Les API Dapr disponibles incluent les appels de service à service, Pub/Sub, les liaisons d’événements, les magasins d’état et les acteurs.
À suivre !




















































Top comments (0)