Dans un article précédent j’avais parlé de l’utilisation d’un client en langage Julia pour Kubernetes via Kuber.jl :
Le langage Julia à l’échelle avec Kubernetes …
Pour rappel, Julia est un langage de programmation récent orienté (très haute) performance : il s’agit de l’un des très rares langages à être utilisé sur un superordinateur.
Julia Computing - Julia Computing
Ici je pars d’un VM Scale Set (groupe de machines virtuelles identiques) dans Azure utilisant de la série NVv4 fonctionnant avec des GPU AMD Radeon Instinct MI25 et des processeurs AMD EPYC 7V12 (Rome) ayant une fréquence de base de 2,45 GHz, une fréquence maximale de 3,1 GHz pour tous les cœurs, et une fréquence maximale de 3,3 GHz pour un seul cœur.
Série NVv4 - Azure Virtual Machines
J’utilise pour plus de facilité au niveau de la connexion à ces instances du client Tailscale :
Accéder à Red Hat CodeReady Containers dans Azure depuis Tailscale …
avec ces deux scripts :
#!/bin/bash
az group create --name RG-JULIA --location eastus
az network nsg create -g RG-JULIA -n nsg-julia
az network vnet create --address-prefixes 10.0.0.0/16 --name Net1 --resource-group RG-JULIA --subnet-name Subnet1 --subnet-prefixes 10.0.0.0/24 --network-security-group nsg-julia
az vmss create \
--resource-group "RG-JULIA" \
--name JULIA \
--instance-count 3 \
--image Canonical:0001-com-ubuntu-server-focal-daily:20_04-daily-lts-gen2:latest \
--vm-sku Standard_NV4as_v4 \
--vnet-name Net1 \
--subnet Subnet1 \
--nsg nsg-julia \
--priority Spot \
--accelerated-networking true \
--lb "" \
--upgrade-policy-mode manual \
--eviction-policy delete \
--admin-username ubuntu \
--ssh-key-values ~/.ssh/id_rsa.pub \
--custom-data ./tailscale-azure.sh
az group delete --resource-group NetworkWatcherRG --yes
et le script de personnalisation suivant pour l’installation de Tailscale :
#!/bin/bash
curl -fsSL [https://pkgs.tailscale.com/stable/ubuntu/focal.gpg](https://pkgs.tailscale.com/stable/ubuntu/focal.gpg) | sudo apt-key add -
curl -fsSL [https://pkgs.tailscale.com/stable/ubuntu/focal.list](https://pkgs.tailscale.com/stable/ubuntu/focal.list) | sudo tee /etc/apt/sources.list.d/tailscale.list
sudo apt-get update -y
sudo apt-get install tailscale -y
sudo tailscale up --advertise-routes=10.0.0.0/24,168.63.129.16/32 --accept-dns=false --authkey XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX
sudo snap remove lxd --purge
echo fs.inotify.max_queued_events=1048576 | sudo tee -a /etc/sysctl.conf
echo fs.inotify.max_user_instances=1048576 | sudo tee -a /etc/sysctl.conf
echo fs.inotify.max_user_watches=1048576 | sudo tee -a /etc/sysctl.conf
echo vm.max_map_count=262144 | sudo tee -a /etc/sysctl.conf
echo vm.swappiness=1 | sudo tee -a /etc/sysctl.conf
sudo umount /dev/sdb1 && sudo mkdir /var/lib/rancher && sudo mount /dev/sdb1 /var/lib/rancher
sudo sysctl -p
Je vois apparaître mes trois instances connectés à Tailscale :
$ tailscale status
100.80.150.62 kvm09e828000000 karim@ linux active;
100.67.185.57 kvm09e828000002 karim@ linux active;
100.89.229.79 kvm09e828000004 karim@ linux active;
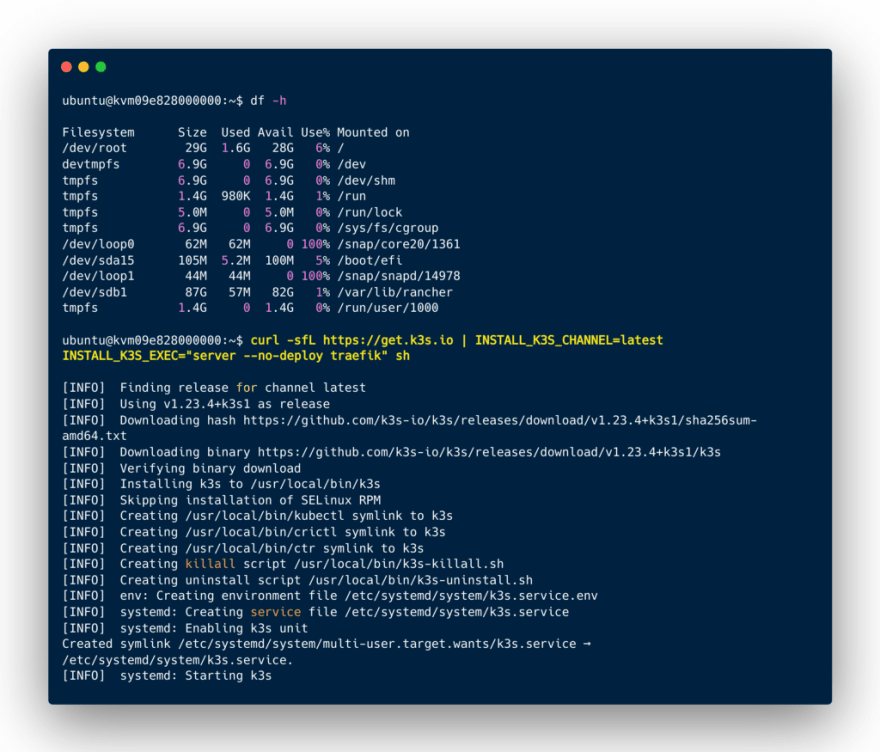
J’installe le noeud maître d’un cluster Kubernetes avec Rancher K3s via ce script sans Traefik :
curl -sfL [https://get.k3s.io](https://get.k3s.io) | INSTALL_K3S_CHANNEL=latest INSTALL_K3S_EXEC="server --no-deploy traefik" s
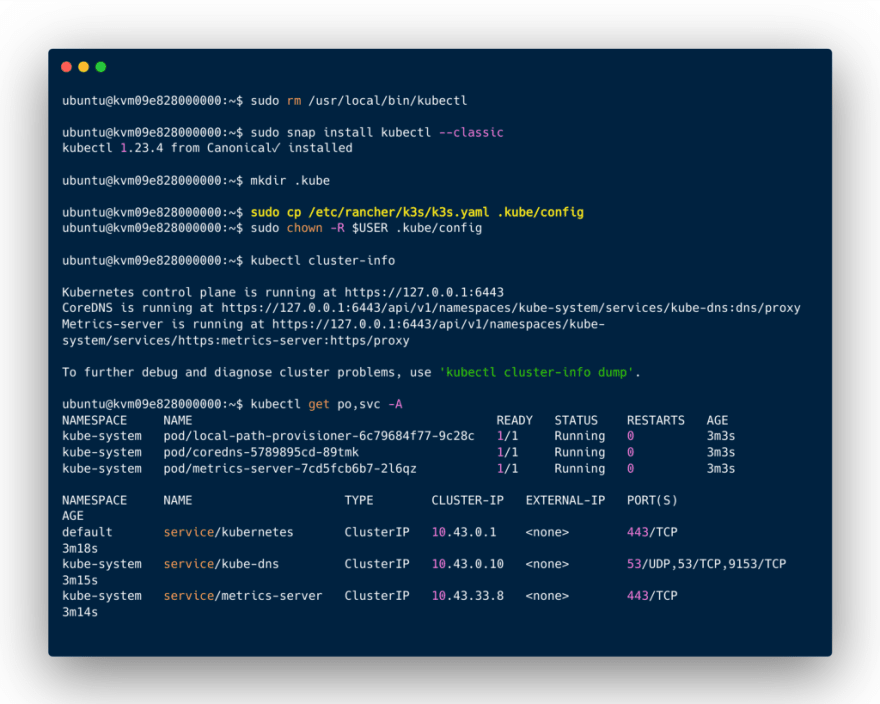
Et je peux interagir avec ce noeud via le client Kubectl :
Je raccorde les deux autres noeuds au cluster :
curl -sfL [https://get.k3s.io](https://get.k3s.io) | INSTALL_K3S_CHANNEL=latest K3S_URL=[https://10.0.0.4:6443](https://10.0.0.4:6443) K3S_TOKEN=K100f55829d124639c99f32d1de45c6a2d7ea131117ea9b8dc739619fa6023f1a27::server:7442e3dfb45532b85e98d6c819dfaa54 sh -
Le cluster Kubernetes est alors constitué avec son noeud maître et ses deux noeuds workers :
Il est donc possible de mettre en oeuvre un gestionnaire de cluster avec Julia pour le provisionnement de workers dans un cluster Kubernetes via K8sClusterManagers.jl :
- K8sClusterManagers.jl
- GitHub - beacon-biosignals/K8sClusterManagers.jl: A Julia cluster manager for Kubernetes
Je commence donc par la création de comptes de services pour les futurs Pod Julia dans ce cluster Kubernetes :
# Minimal set of permissions required by K8sClusterManagers.jl
apiVersion: rbac.authorization.k8s.io/v1
kind: Role
metadata:
name: julia-manager-role
rules:
- apiGroups: [""] # "" indicates the core API group
resources: ["pods"]
verbs: ["create", "delete", "get", "patch"]
- apiGroups: [""]
resources: ["pods/exec"]
verbs: ["create"]
- apiGroups: [""]
resources: ["pods/log"]
verbs: ["get"]
---
# https://kubernetes.io/docs/tasks/configure-pod-container/configure-service-account/
apiVersion: v1
kind: ServiceAccount
metadata:
name: julia-manager-serviceaccount
automountServiceAccountToken: true
---
apiVersion: rbac.authorization.k8s.io/v1
kind: RoleBinding
metadata:
name: julia-manager-role-binding
roleRef:
kind: Role
name: julia-manager-role
apiGroup: rbac.authorization.k8s.io
subjects:
- kind: ServiceAccount
name: julia-manager-serviceaccount
ubuntu@kvm09e828000000:~$ kubectl apply -f [https://raw.githubusercontent.com/beacon-biosignals/K8sClusterManagers.jl/main/docs/src/julia-manager-serviceaccount.yaml](https://raw.githubusercontent.com/beacon-biosignals/K8sClusterManagers.jl/main/docs/src/julia-manager-serviceaccount.yaml)
role.rbac.authorization.k8s.io/julia-manager-role created
serviceaccount/julia-manager-serviceaccount created
rolebinding.rbac.authorization.k8s.io/julia-manager-role-binding created
Lancement du premier Pod avec la langage Julia via ce manifest YAML :
apiVersion: apps/v1
kind: Deployment
metadata:
creationTimestamp: null
labels:
app: julia
name: julia
spec:
replicas: 1
selector:
matchLabels:
app: julia
strategy: {}
template:
metadata:
creationTimestamp: null
labels:
app: julia
spec:
serviceAccountName: julia-manager-serviceaccount
containers:
- image: [julia:latest](http://docker.io/library/julia:latest)
name: julia
stdin: true
tty: true
command: ["/bin/sleep", "3650d"]
resources: {}
---
apiVersion: v1
kind: Service
metadata:
name: julia
spec:
ports:
- name: julia-service
port: 80
protocol: TCP
targetPort: 1234
selector:
app: julia
type: LoadBalancer
Je lance un shell dans le Pod avec Julia :
ubuntu@kvm09e828000000:~$ kubectl exec -it pod/julia-75444d5c79-686cf -- /bin/bash
root@julia-75444d5c79-686cf:/# apt install screen -y
Installation de Pyston, une distribution Python pour installer Jupyter Lab via Conda …
root@julia-75444d5c79-686cf:/# curl -LO [https://github.com/pyston/pyston/releases/download/pyston\_2.3.2/PystonConda-1.1-Linux-x86\_64.sh](https://github.com/pyston/pyston/releases/download/pyston_2.3.2/PystonConda-1.1-Linux-x86_64.sh)
% Total % Received % Xferd Average Speed Time Time Time Current
Dload Upload Total Spent Left Speed
100 670 100 670 0 0 8072 0 --:--:-- --:--:-- --:--:-- 7976
100 88.2M 100 88.2M 0 0 89.3M 0 --:--:-- --:--:-- --:--:-- 89.3M
root@julia-75444d5c79-686cf:/# chmod +x PystonConda-1.1-Linux-x86_64.sh
root@julia-75444d5c79-686cf:/# ./PystonConda-1.1-Linux-x86_64.sh
Welcome to PystonConda 1.1
In order to continue the installation process, please review the license
agreement.
Please, press ENTER to continue
>>>
PystonConda installer code uses BSD-3-Clause license as stated below.
Binary packages that come with it have their own licensing terms
and by installing PystonConda you agree to the licensing terms of individual
packages as well. They include different OSI-approved licenses including
the GNU General Public License and can be found in pkgs/<pkg-name>/info/licenses
folders.
=============================================================================
Copyright (c) 2021, Anaconda, Inc.
All rights reserved.
Redistribution and use in source and binary forms, with or without
modification, are permitted provided that the following conditions are met:
* Redistributions of source code must retain the above copyright
notice, this list of conditions and the following disclaimer.
* Redistributions in binary form must reproduce the above copyright
notice, this list of conditions and the following disclaimer in the
documentation and/or other materials provided with the distribution.
* Neither the name of Anaconda, Inc. nor the names of its contributors
may be used to endorse or promote products derived from this software
without specific prior written permission.
THIS SOFTWARE IS PROVIDED BY THE COPYRIGHT HOLDERS AND CONTRIBUTORS "AS IS" AND
ANY EXPRESS OR IMPLIED WARRANTIES, INCLUDING, BUT NOT LIMITED TO, THE IMPLIED
WARRANTIES OF MERCHANTABILITY AND FITNESS FOR A PARTICULAR PURPOSE ARE
DISCLAIMED. IN NO EVENT SHALL ANACONDA, INC BE LIABLE FOR ANY
DIRECT, INDIRECT, INCIDENTAL, SPECIAL, EXEMPLARY, OR CONSEQUENTIAL DAMAGES
(INCLUDING, BUT NOT LIMITED TO, PROCUREMENT OF SUBSTITUTE GOODS OR SERVICES;
LOSS OF USE, DATA, OR PROFITS; OR BUSINESS INTERRUPTION) HOWEVER CAUSED AND
ON ANY THEORY OF LIABILITY, WHETHER IN CONTRACT, STRICT LIABILITY, OR TORT
(INCLUDING NEGLIGENCE OR OTHERWISE) ARISING IN ANY WAY OUT OF THE USE OF THIS
SOFTWARE, EVEN IF ADVISED OF THE POSSIBILITY OF SUCH DAMAGE.
Do you accept the license terms? [yes|no]
[no] >>> yes
PystonConda will now be installed into this location:
/root/pystonconda
- Press ENTER to confirm the location
- Press CTRL-C to abort the installation
- Or specify a different location below
[/root/pystonconda] >>>
PREFIX=/root/pystonconda
Unpacking payload ...
Collecting package metadata (current_repodata.json): done
Solving environment: done
## Package Plan ##
environment location: /root/pystonconda
added / updated specs:
- _libgcc_mutex==0.1=conda_forge
- _openmp_mutex==4.5=1_gnu
- brotlipy==0.7.0=py38h79d3a15_1003
- bzip2==1.0.8=h7f98852_4
- ca-certificates==2021.10.8=ha878542_0
- certifi==2021.10.8=py38hc2d5299_1
- cffi==1.15.0=py38h9a12ab7_0
- charset-normalizer==2.0.11=pyhd8ed1ab_0
- colorama==0.4.4=pyh9f0ad1d_0
- conda-package-handling==1.7.3=py38h79d3a15_1
- conda==4.11.0=py38h4c12d10_0
- cryptography==36.0.0=py38ha252339_0
- freetype==2.10.4=h0708190_1
- idna==3.3=pyhd8ed1ab_0
- jbig==2.1=h7f98852_2003
- jpeg==9e=h7f98852_0
- lerc==3.0=h9c3ff4c_0
- libdeflate==1.8=h7f98852_0
- libffi==3.4.2=h7f98852_5
- libgcc-ng==11.2.0=h1d223b6_12
- libgomp==11.2.0=h1d223b6_12
- libpng==1.6.37=h21135ba_2
- libstdcxx-ng==11.2.0=he4da1e4_12
- libtiff==4.3.0=h6f004c6_2
- libwebp-base==1.2.2=h7f98852_1
- libzlib==1.2.11=h36c2ea0_1013
- lz4-c==1.9.3=h9c3ff4c_1
- ncurses==6.2=h58526e2_4
- openssl==1.1.1l=h7f98852_0
- pip==22.0.3=pyhd8ed1ab_0
- pycosat==0.6.3=py38h79d3a15_1009
- pycparser==2.21=pyhd8ed1ab_0
- pyopenssl==22.0.0=pyhd8ed1ab_0
- pysocks==1.7.1=py38h4c12d10_4
- pyston2.3==2.3.2=0_23_pyston
- pyston==2.3.2=3
- python==3.8.12=3_23_pyston
- python_abi==3.8=1_23_pyston
- readline==8.1=h46c0cb4_0
- requests==2.27.1=pyhd8ed1ab_0
- ruamel_yaml==0.15.80=py38h79d3a15_1006
- setuptools==60.7.0=py38hc2d5299_0
- six==1.16.0=pyh6c4a22f_0
- sqlite==3.37.0=h9cd32fc_0
- tk==8.6.11=h27826a3_1
- tqdm==4.62.3=pyhd8ed1ab_0
- tzdata==2021e=he74cb21_0
- urllib3==1.26.8=pyhd8ed1ab_1
- wheel==0.37.1=pyhd8ed1ab_0
- xz==5.2.5=h516909a_1
- yaml==0.2.5=h7f98852_2
- zlib==1.2.11=h36c2ea0_1013
- zstd==1.5.2=ha95c52a_0
The following NEW packages will be INSTALLED:
_libgcc_mutex conda-forge/linux-64::_libgcc_mutex-0.1-conda_forge
_openmp_mutex conda-forge/linux-64::_openmp_mutex-4.5-1_gnu
brotlipy pyston/linux-64::brotlipy-0.7.0-py38h79d3a15_1003
bzip2 conda-forge/linux-64::bzip2-1.0.8-h7f98852_4
ca-certificates conda-forge/linux-64::ca-certificates-2021.10.8-ha878542_0
certifi pyston/linux-64::certifi-2021.10.8-py38hc2d5299_1
cffi pyston/linux-64::cffi-1.15.0-py38h9a12ab7_0
charset-normalizer conda-forge/noarch::charset-normalizer-2.0.11-pyhd8ed1ab_0
colorama conda-forge/noarch::colorama-0.4.4-pyh9f0ad1d_0
conda pyston/linux-64::conda-4.11.0-py38h4c12d10_0
conda-package-han~ pyston/linux-64::conda-package-handling-1.7.3-py38h79d3a15_1
cryptography pyston/linux-64::cryptography-36.0.0-py38ha252339_0
freetype conda-forge/linux-64::freetype-2.10.4-h0708190_1
idna conda-forge/noarch::idna-3.3-pyhd8ed1ab_0
jbig conda-forge/linux-64::jbig-2.1-h7f98852_2003
jpeg conda-forge/linux-64::jpeg-9e-h7f98852_0
lerc conda-forge/linux-64::lerc-3.0-h9c3ff4c_0
libdeflate conda-forge/linux-64::libdeflate-1.8-h7f98852_0
libffi conda-forge/linux-64::libffi-3.4.2-h7f98852_5
libgcc-ng conda-forge/linux-64::libgcc-ng-11.2.0-h1d223b6_12
libgomp conda-forge/linux-64::libgomp-11.2.0-h1d223b6_12
libpng conda-forge/linux-64::libpng-1.6.37-h21135ba_2
libstdcxx-ng conda-forge/linux-64::libstdcxx-ng-11.2.0-he4da1e4_12
libtiff conda-forge/linux-64::libtiff-4.3.0-h6f004c6_2
libwebp-base conda-forge/linux-64::libwebp-base-1.2.2-h7f98852_1
libzlib conda-forge/linux-64::libzlib-1.2.11-h36c2ea0_1013
lz4-c conda-forge/linux-64::lz4-c-1.9.3-h9c3ff4c_1
ncurses conda-forge/linux-64::ncurses-6.2-h58526e2_4
openssl conda-forge/linux-64::openssl-1.1.1l-h7f98852_0
pip conda-forge/noarch::pip-22.0.3-pyhd8ed1ab_0
pycosat pyston/linux-64::pycosat-0.6.3-py38h79d3a15_1009
pycparser conda-forge/noarch::pycparser-2.21-pyhd8ed1ab_0
pyopenssl conda-forge/noarch::pyopenssl-22.0.0-pyhd8ed1ab_0
pysocks pyston/linux-64::pysocks-1.7.1-py38h4c12d10_4
pyston pyston/noarch::pyston-2.3.2-3
pyston2.3 pyston/linux-64::pyston2.3-2.3.2-0_23_pyston
python pyston/linux-64::python-3.8.12-3_23_pyston
python_abi pyston/linux-64::python_abi-3.8-1_23_pyston
readline conda-forge/linux-64::readline-8.1-h46c0cb4_0
requests conda-forge/noarch::requests-2.27.1-pyhd8ed1ab_0
ruamel_yaml pyston/linux-64::ruamel_yaml-0.15.80-py38h79d3a15_1006
setuptools pyston/linux-64::setuptools-60.7.0-py38hc2d5299_0
six conda-forge/noarch::six-1.16.0-pyh6c4a22f_0
sqlite conda-forge/linux-64::sqlite-3.37.0-h9cd32fc_0
tk conda-forge/linux-64::tk-8.6.11-h27826a3_1
tqdm conda-forge/noarch::tqdm-4.62.3-pyhd8ed1ab_0
tzdata conda-forge/noarch::tzdata-2021e-he74cb21_0
urllib3 conda-forge/noarch::urllib3-1.26.8-pyhd8ed1ab_1
wheel conda-forge/noarch::wheel-0.37.1-pyhd8ed1ab_0
xz conda-forge/linux-64::xz-5.2.5-h516909a_1
yaml conda-forge/linux-64::yaml-0.2.5-h7f98852_2
zlib conda-forge/linux-64::zlib-1.2.11-h36c2ea0_1013
zstd conda-forge/linux-64::zstd-1.5.2-ha95c52a_0
Preparing transaction: done
Executing transaction: done
installation finished.
Do you wish the installer to initialize PystonConda
by running conda init? [yes|no]
[no] >>> yes
no change /root/pystonconda/condabin/conda
no change /root/pystonconda/bin/conda
no change /root/pystonconda/bin/conda-env
no change /root/pystonconda/bin/activate
no change /root/pystonconda/bin/deactivate
no change /root/pystonconda/etc/profile.d/conda.sh
no change /root/pystonconda/etc/fish/conf.d/conda.fish
no change /root/pystonconda/shell/condabin/Conda.psm1
no change /root/pystonconda/shell/condabin/conda-hook.ps1
no change /root/pystonconda/lib/python3.8/site-packages/xontrib/conda.xsh
no change /root/pystonconda/etc/profile.d/conda.csh
modified /root/.bashrc
==> For changes to take effect, close and re-open your current shell. <==
If you'd prefer that conda's base environment not be activated on startup,
set the auto_activate_base parameter to false:
conda config --set auto_activate_base false
Thank you for installing PystonConda!
Installation - JupyterLab 3.3.0 documentation
(base) root@julia-75444d5c79-686cf:/# conda install -c conda-forge jupyterlab
Collecting package metadata (current_repodata.json): done
Solving environment: done
## Package Plan ##
environment location: /root/pystonconda
added / updated specs:
- jupyterlab
The following packages will be downloaded:
package | build
---------------------------|-----------------
anyio-3.4.0 | py38h4c12d10_0 150 KB pyston
argon2-cffi-21.1.0 | py38h79d3a15_2 46 KB pyston
attrs-21.4.0 | pyhd8ed1ab_0 49 KB conda-forge
babel-2.9.1 | pyh44b312d_0 6.2 MB conda-forge
backcall-0.2.0 | pyh9f0ad1d_0 13 KB conda-forge
backports-1.0 | py_2 4 KB conda-forge
backports.functools_lru_cache-1.6.4| pyhd8ed1ab_0 9 KB conda-forge
bleach-4.1.0 | pyhd8ed1ab_0 121 KB conda-forge
debugpy-1.5.1 | py38hec7152c_0 2.0 MB pyston
decorator-5.1.1 | pyhd8ed1ab_0 12 KB conda-forge
defusedxml-0.7.1 | pyhd8ed1ab_0 23 KB conda-forge
entrypoints-0.4 | pyhd8ed1ab_0 9 KB conda-forge
importlib-metadata-3.3.0 | pyhd8ed1ab_1 12 KB conda-forge
importlib_resources-5.4.0 | pyhd8ed1ab_0 21 KB conda-forge
ipykernel-6.5.0 | py38h8805fdd_1 177 KB pyston
ipython-7.30.0 | py38h4c12d10_0 1.1 MB pyston
ipython_genutils-0.2.0 | py_1 21 KB conda-forge
jedi-0.18.1 | py38h4c12d10_0 998 KB pyston
jinja2-3.0.3 | pyhd8ed1ab_0 99 KB conda-forge
json5-0.9.5 | pyh9f0ad1d_0 20 KB conda-forge
jsonschema-4.4.0 | pyhd8ed1ab_0 57 KB conda-forge
jupyter_client-7.1.2 | pyhd8ed1ab_0 90 KB conda-forge
jupyter_core-4.9.1 | py38h4c12d10_1 80 KB pyston
jupyter_server-1.13.5 | pyhd8ed1ab_1 261 KB conda-forge
jupyterlab-3.3.1 | pyhd8ed1ab_0 5.9 MB conda-forge
jupyterlab_server-2.10.3 | pyhd8ed1ab_0 47 KB conda-forge
libsodium-1.0.18 | h36c2ea0_1 366 KB conda-forge
markupsafe-2.0.1 | py38h79d3a15_1 22 KB pyston
matplotlib-inline-0.1.3 | pyhd8ed1ab_0 11 KB conda-forge
mistune-0.8.3 | py_0 16 KB conda-forge
nbclassic-0.3.6 | pyhd8ed1ab_0 15 KB conda-forge
nbconvert-5.6.1 | pyhd8ed1ab_2 373 KB conda-forge
nbformat-5.2.0 | pyhd8ed1ab_0 105 KB conda-forge
nest-asyncio-1.5.4 | pyhd8ed1ab_0 9 KB conda-forge
notebook-6.4.8 | pyha770c72_0 6.3 MB conda-forge
notebook-shim-0.1.0 | pyhd8ed1ab_0 15 KB conda-forge
packaging-21.3 | pyhd8ed1ab_0 36 KB conda-forge
pandocfilters-1.5.0 | pyhd8ed1ab_0 11 KB conda-forge
parso-0.8.3 | pyhd8ed1ab_0 69 KB conda-forge
pexpect-4.8.0 | pyh9f0ad1d_2 47 KB conda-forge
pickleshare-0.7.5 | py_1003 9 KB conda-forge
prometheus_client-0.13.1 | pyhd8ed1ab_0 47 KB conda-forge
prompt-toolkit-3.0.27 | pyha770c72_0 252 KB conda-forge
ptyprocess-0.7.0 | pyhd3deb0d_0 16 KB conda-forge
pygments-2.11.2 | pyhd8ed1ab_0 796 KB conda-forge
pyparsing-3.0.7 | pyhd8ed1ab_0 79 KB conda-forge
pyrsistent-0.18.0 | py38h79d3a15_0 91 KB pyston
python-dateutil-2.8.2 | pyhd8ed1ab_0 240 KB conda-forge
pytz-2021.3 | pyhd8ed1ab_0 242 KB conda-forge
pyzmq-22.3.0 | py38hcdae430_1 527 KB pyston
send2trash-1.8.0 | pyhd8ed1ab_0 17 KB conda-forge
sniffio-1.2.0 | py38h4c12d10_2 16 KB pyston
terminado-0.12.1 | py38h4c12d10_1 28 KB pyston
testpath-0.6.0 | pyhd8ed1ab_0 85 KB conda-forge
tornado-6.1 | py38h79d3a15_2 650 KB pyston
traitlets-5.1.1 | pyhd8ed1ab_0 82 KB conda-forge
typing_extensions-4.1.1 | pyha770c72_0 29 KB conda-forge
wcwidth-0.2.5 | pyh9f0ad1d_2 33 KB conda-forge
webencodings-0.5.1 | py_1 12 KB conda-forge
websocket-client-1.3.1 | pyhd8ed1ab_0 41 KB conda-forge
zeromq-4.3.4 | h9c3ff4c_1 351 KB conda-forge
zipp-3.7.0 | pyhd8ed1ab_1 12 KB conda-forge
------------------------------------------------------------
Total: 28.4 MB
Installation du Kernel IJulia et lancement de Jupyter Lab :
GitHub - JuliaLang/IJulia.jl: Julia kernel for Jupyter
(base) root@julia-75444d5c79-686cf:/# julia
_
_ _ _(_)_ | Documentation: [https://docs.julialang.org](https://docs.julialang.org)
(_) | (_) (_) |
_ _ _| |_ __ _ | Type "?" for help, "]?" for Pkg help.
| | | | | | |/ _` | |
| | |_| | | | (_| | | Version 1.7.2 (2022-02-06)
_/ |\ __'_|_|_|\__'_| | Official [https://julialang.org/](https://julialang.org/) release
|__/ |
([@v1](http://twitter.com/v1).7) pkg> add IJulia
Installing known registries into `~/.julia`
Updating registry at `~/.julia/registries/General.toml`
Resolving package versions...
Installed VersionParsing ── v1.3.0
Installed Conda ─────────── v1.7.0
Installed Parsers ───────── v2.2.3
Installed Preferences ───── v1.2.5
Installed ZMQ ───────────── v1.2.1
Installed IJulia ────────── v1.23.2
Installed JSON ──────────── v0.21.3
Installed JLLWrappers ───── v1.4.1
Installed ZeroMQ_jll ────── v4.3.4+0
Installed SoftGlobalScope ─ v1.1.0
Installed libsodium_jll ─── v1.0.20+0
Installed MbedTLS ───────── v1.0.3
Downloaded artifact: ZeroMQ
Downloaded artifact: libsodium
Updating `~/.julia/environments/v1.7/Project.toml`
[7073ff75] + IJulia v1.23.2
Updating `~/.julia/environments/v1.7/Manifest.toml`
[8f4d0f93] + Conda v1.7.0
[7073ff75] + IJulia v1.23.2
[692b3bcd] + JLLWrappers v1.4.1
[682c06a0] + JSON v0.21.3
[739be429] + MbedTLS v1.0.3
[69de0a69] + Parsers v2.2.3
[21216c6a] + Preferences v1.2.5
[b85f4697] + SoftGlobalScope v1.1.0
[81def892] + VersionParsing v1.3.0
[c2297ded] + ZMQ v1.2.1
[0dad84c5] + ArgTools
[56f22d72] + Artifacts
[2a0f44e3] + Base64
[ade2ca70] + Dates
[f43a241f] + Downloads
[7b1f6079] + FileWatching
[b77e0a4c] + InteractiveUtils
[b27032c2] + LibCURL
[76f85450] + LibGit2
[8f399da3] + Libdl
[56ddb016] + Logging
[d6f4376e] + Markdown
[a63ad114] + Mmap
[ca575930] + NetworkOptions
[44cfe95a] + Pkg
[de0858da] + Printf
[3fa0cd96] + REPL
[9a3f8284] + Random
[ea8e919c] + SHA
[9e88b42a] + Serialization
[6462fe0b] + Sockets
[fa267f1f] + TOML
[a4e569a6] + Tar
[8dfed614] + Test
[cf7118a7] + UUIDs
[4ec0a83e] + Unicode
[deac9b47] + LibCURL_jll
[29816b5a] + LibSSH2_jll
[c8ffd9c3] + MbedTLS_jll
[14a3606d] + MozillaCACerts_jll
[83775a58] + Zlib_jll
[8e850ede] + nghttp2_jll
[3f19e933] + p7zip_jll
Building Conda ─→ `~/.julia/scratchspaces/44cfe95a-1eb2-52ea-b672-e2afdf69b78f/6e47d11ea2776bc5627421d59cdcc1296c058071/build.log`
Building IJulia → `~/.julia/scratchspaces/44cfe95a-1eb2-52ea-b672-e2afdf69b78f/d8b9c31196e1dd92181cd0f5760ca2d2ffb4ac0f/build.log`
Progress [====================>] 1/2
Precompiling project...
11 dependencies successfully precompiled in 4 seconds (4 already precompiled)
(base) root@julia-75444d5c79-686cf:/# screen -L -S julia
(base) root@julia-75444d5c79-686cf:/# cat screenlog.0
# bash
(base) root@julia-75444d5c79-686cf:/# jupyter lab --ip "0.0.0.0" --port "1234" --allow-root
[I 2022-03-12 19:56:19.308 ServerApp] jupyterlab | extension was successfully linked.
[I 2022-03-12 19:56:19.314 ServerApp] nbclassic | extension was successfully linked.
[I 2022-03-12 19:56:19.315 ServerApp] Writing Jupyter server cookie secret to /root/.local/share/jupyter/runtime/jupyter_cookie_secret
[I 2022-03-12 19:56:19.417 ServerApp] notebook_shim | extension was successfully linked.
[I 2022-03-12 19:56:19.465 ServerApp] notebook_shim | extension was successfully loaded.
[I 2022-03-12 19:56:19.465 LabApp] JupyterLab extension loaded from /root/pystonconda/lib/python3.8-pyston2.3/site-packages/jupyterlab
[I 2022-03-12 19:56:19.465 LabApp] JupyterLab application directory is /root/pystonconda/share/jupyter/lab
[I 2022-03-12 19:56:19.467 ServerApp] jupyterlab | extension was successfully loaded.
[I 2022-03-12 19:56:19.469 ServerApp] nbclassic | extension was successfully loaded.
[I 2022-03-12 19:56:19.469 ServerApp] Serving notebooks from local directory: /
[I 2022-03-12 19:56:19.469 ServerApp] Jupyter Server 1.13.5 is running at:
[I 2022-03-12 19:56:19.469 ServerApp] [http://julia-75444d5c79-686cf:1234/lab?token=c87f67a6e002957c2e49032e02fb81951ec8a569f75c6fc2](http://julia-75444d5c79-686cf:1234/lab?token=c87f67a6e002957c2e49032e02fb81951ec8a569f75c6fc2)
[I 2022-03-12 19:56:19.470 ServerApp] or [http://127.0.0.1:1234/lab?token=c87f67a6e002957c2e49032e02fb81951ec8a569f75c6fc2](http://127.0.0.1:1234/lab?token=c87f67a6e002957c2e49032e02fb81951ec8a569f75c6fc2)
[I 2022-03-12 19:56:19.470 ServerApp] Use Control-C to stop this server and shut down all kernels (twice to skip confirmation).
[W 2022-03-12 19:56:19.471 ServerApp] No web browser found: could not locate runnable browser.
[C 2022-03-12 19:56:19.472 ServerApp]
To access the server, open this file in a browser:
file:///root/.local/share/jupyter/runtime/jpserver-1096-open.html
Or copy and paste one of these URLs:
[http://julia-75444d5c79-686cf:1234/lab?token=c87f67a6e002957c2e49032e02fb81951ec8a569f75c6fc2](http://julia-75444d5c79-686cf:1234/lab?token=c87f67a6e002957c2e49032e02fb81951ec8a569f75c6fc2)
or [http://127.0.0.1:1234/lab?token=c87f67a6e002957c2e49032e02fb81951ec8a569f75c6fc2](http://127.0.0.1:1234/lab?token=c87f67a6e002957c2e49032e02fb81951ec8a569f75c6fc2)
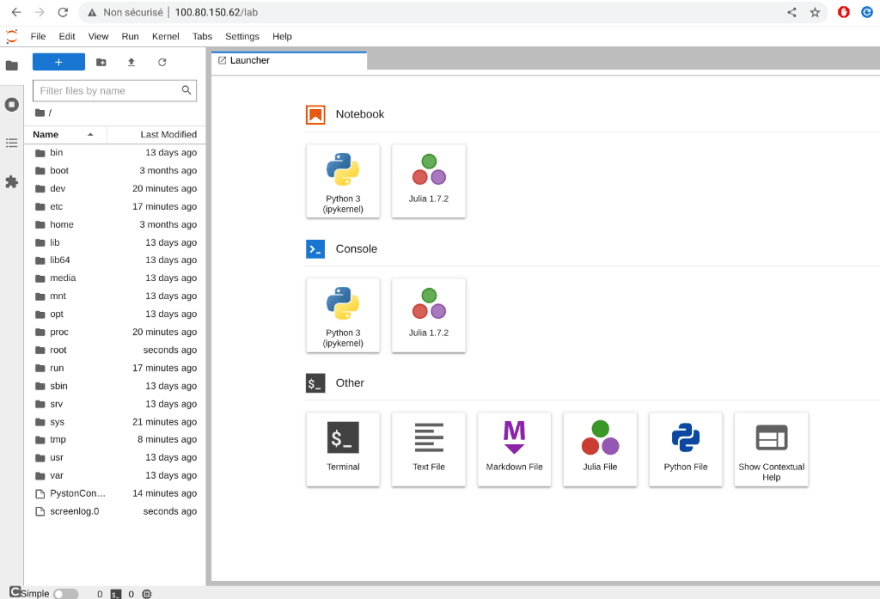
Comme le cluster K3s est installé sans Traefik, je peux beneficier du service LoadBalancer via Klipper, ce qui me permet d’accéder à l’interface de Jupyter Lab sur le port HTTP/80 via l’une des adresses IP fournies par Tailscale :
GitHub - k3s-io/klipper-lb: Embedded service load balancer in Klipper
ubuntu@kvm09e828000000:~$ kubectl get svc -A
NAMESPACE NAME TYPE CLUSTER-IP EXTERNAL-IP PORT(S) AGE
default kubernetes ClusterIP 10.43.0.1 <none> 443/TCP 44m
kube-system kube-dns ClusterIP 10.43.0.10 <none> 53/UDP,53/TCP,9153/TCP 44m
kube-system metrics-server ClusterIP 10.43.33.8 <none> 443/TCP 44m
default julia LoadBalancer 10.43.52.50 10.0.0.4,10.0.0.6,10.0.0.8 80:31333/TCP 17m
K8sClusterManagers est mis en oeuvre ici pour permettre le lancement des futurs workers via Jupyter Lab et le notebook Julia :
using Pkg
import Pkg
Pkg.add("K8sClusterManagers")
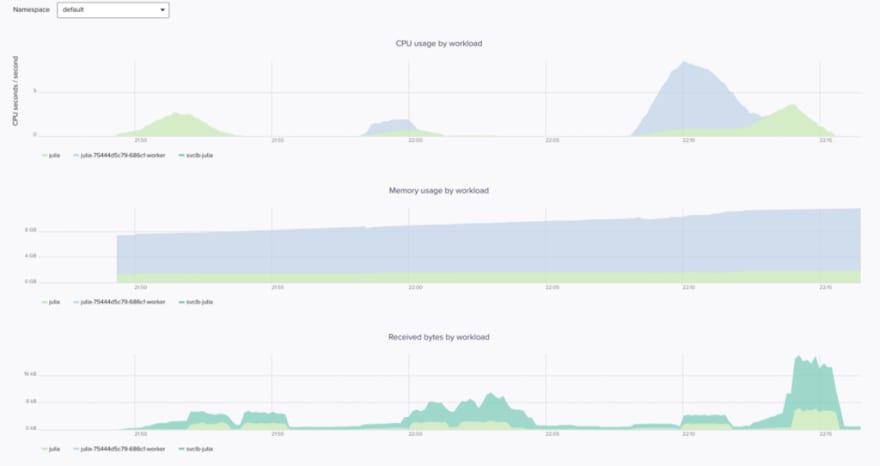
Les workers Julia, ici au nombre de 8, peuvent être lancés toujours via le notebook :
using Distributed, K8sClusterManagers
addprocs(K8sClusterManager(8))
Les Pods sont alors visibles et en exécution notamment via Weave Scope :
Notre compréhension des phénomènes du monde réel et notre technologie sont aujourd’hui en grande partie basées sur les équations aux dérivées partielles ou EDP.
Équation aux dérivées partielles - Wikipédia
J’utilise ici NeuralPDE.jl, un paquet de solveurs qui consiste en des solveurs de réseaux neuronaux pour les équations aux dérivées partielles utilisant du Scientific Machine Learning (SciML) telles que les réseaux neuronaux informés par la physique (PINN) et les solveurs BSDE profonds.
Ce paquetage utilise des réseaux neuronaux profonds et des équations différentielles stochastiques neuronales pour résoudre des EDP de haute dimension à un coût considérablement réduit et avec une généralité considérablement accrue par rapport aux méthodes classiques.
- GitHub - SciML/NeuralPDE.jl: Physics-Informed Neural Networks (PINN) and Deep BSDE Solvers of Differential Equations for Scientific Machine Learning (SciML) accelerated simulation
- SciML Open Source Scientific Machine Learning
- SciML
Je vais me baser pour l’utilisation de ces Pods Julia, des exemples proposés par NeuralPDE.jl :
NeuralPDE.jl: Scientific Machine Learning for Partial Differential Equations
et ce dépôt pour bénéficier du mode distribué :
GitHub - juliohm/julia-distributed-computing: The ultimate guide to distributed computing in Julia
Les réseaux neuronaux à information physique (PINN) constituent un moyen de plus en plus puissant de résoudre des équations aux dérivées partielles, de générer des jumeaux numériques et de créer des substituts neuronaux de modèles physiques. Dans cet article on détaille le fonctionnement interne de NeuralPDE.jl afin d’aider les utilisateurs potentiels à adopter ces techniques basées sur les PINN.
NeuralPDE: Automating Physics-Informed Neural Networks (PINNs) with Error Approximations
NeuralPDE.jl utilise une formulation purement symbolique de sorte que tout le code d’entraînement sous-jacent est généré à partir d’une formulation abstraite. Cela permet l’utilisation de réseaux neuronaux basés sur la physique pour la résolution automatisée des EDP, des méthodes FBSDE (Forward-Backwards Stochastic Differential Equation) pour les EDP paraboliques. Ainsi que des solveurs basés sur l’apprentissage profond pour le temps d’arrêt optimal et les équations de Kolmogorov à rebours.
NeuralPDE.jl: Scientific Machine Learning for Partial Differential Equations
Installation des dépendances nécessaires en Julia pour bénéficier des réseaux neuronaux informés par la physique ou PINN (physics-informed neural networks) :
Physics-informed neural networks - Wikipedia
@everywhere begin
using Pkg
dependencies = [
"Plots",
"NeuralPDE",
"Flux",
"ModelingToolkit",
"GalacticOptim",
"Optim",
"DiffEqFlux",
"Quadrature",
"Cubature"
]
Pkg.add(dependencies)
Pkg.instantiate()
end
Dans cet exemple, on va résoudre le système d’équations aux dérivées partielles suivant :
avec ces conditions initiales :
et ces conditions aux limites :
avec des réseaux neuronaux basés sur la physique via NeuralPDE.jl :
Je peux alors visualiser quelques analyses :
Autre exemple en prenant l’équation de Burgers 1-D. L’équation de Burgers ou équation de Bateman-Burgers est une équation différentielle partielle fondamentale apparaissant dans divers domaines des mathématiques appliquées, tels que la mécanique des fluides, l’acoustique non linéaire, la dynamique des gaz et l’écoulement du trafic. L’équation a été introduite pour la première fois par Harry Bateman en 1915, puis étudiée par Johannes Martinus Burgers en 1948 :
avec quelques analyses :
et le monitoring via Weave Cloud :
Considérons l’équation Kuramoto-Sivashinsky, qui contient une dérivée d’ordre 4.
En mathématiques, l’équation de Kuramoto-Sivashinsky (également appelée équation KS ou équation de la flamme) est une équation différentielle partielle non linéaire d’ordre 4. Elle porte le nom de Yoshiki Kuramoto et Gregory Sivashinsky, qui ont dérivé l’équation à la fin des années 1970 pour modéliser les instabilités diffusives dans un front de flamme laminaire . L’équation Kuramoto-Sivashinsky est connue pour son comportement chaotique :
Kuramoto-Sivashinsky equation - Wikipedia
où alpha = gamma = 1 et beta = 4. La solution exacte est :
où thêta = 1-x/2 et avec les conditions initiales et limites :
J’utilise encore dans cet exemple des réseaux de neurones informés par la physique :
Et quelques solutions :
Exemple avec l’équation des ondes 1D avec conditions aux limites de Dirichlet.
En mathématiques, l’équation de d’Alembert (parfois appelée équation d’onde ou équation des ondes) est l’équation générale qui décrit la propagation d’une onde, qui peut être représentée par une grandeur scalaire ou vectorielle. Alors qu’une condition aux limites de Dirichlet (nommée d’après Johann Dirichlet) est imposée à une équation différentielle ou à une équation aux dérivées partielles lorsque l’on spécifie les valeurs que la solution doit vérifier sur les frontières/limites du domaine :
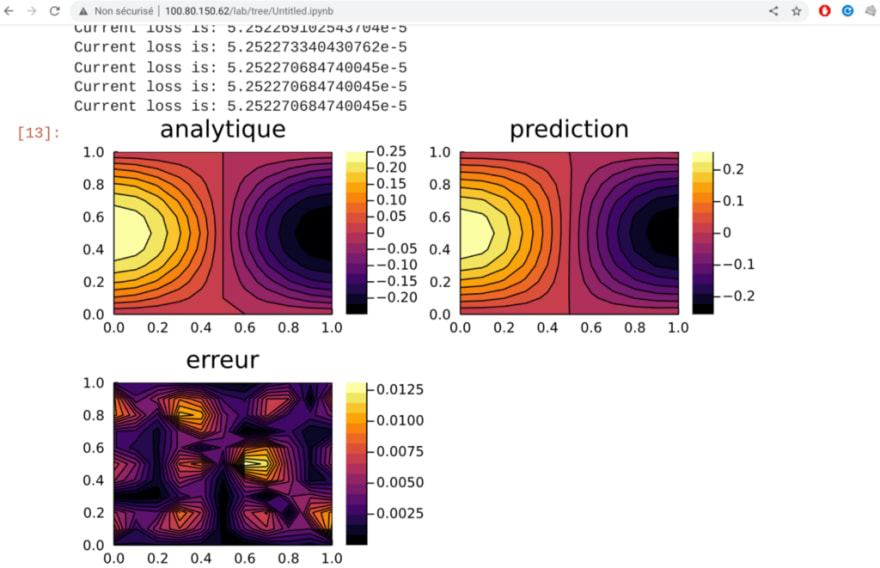
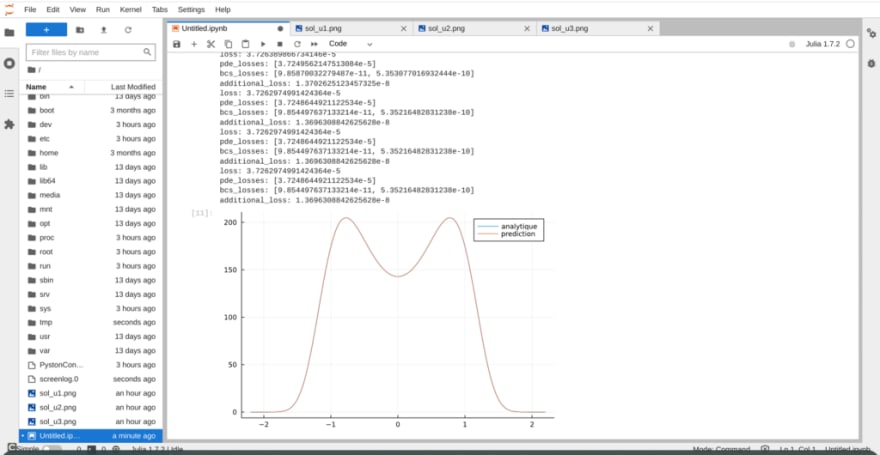
Résolution de cette équation d’onde unidimensionnelle,
avec une discrétisation par grille dx = 0,1 et des réseaux neuronaux informés par la physique. La solution de cette équation avec les conditions aux limites données est présentée via ce notebook :
On peut alors tracer la solution prédite de l’EDP et la comparer avec la solution analytique afin de tracer l’erreur relative :
Je pars du dernier exemple avec l’équation de Fokker-Planck, une équation aux dérivées partielles linéaire que doit satisfaire la densité de probabilité de transition d’un processus de Markov.
À l’origine, une forme simplifiée de cette équation a permis d’étudier le mouvement brownien. Comme la plupart des équations aux dérivées partielles, elle ne donne des solutions explicites que dans des cas bien particuliers portant à la fois sur la forme de l’équation, sur la forme du domaine où elle est étudiée (conditions réfléchissante ou absorbante pour les particules browniennes et forme de l’espace dans lequel elles sont confinées par exemple). Elle est nommée en l’honneur d’Adriaan Fokker et de Max Planck, les premiers physiciens à l’avoir proposée.
Fokker-Planck equation - Wikipedia
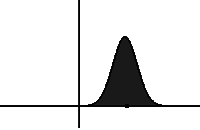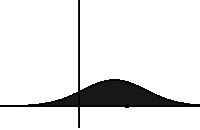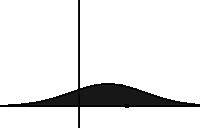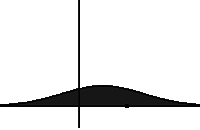
Une solution à l’équation de Fokker-Planck unidimensionnelle, avec les deux termes de dérive et de diffusion.Au fil du temps, la distribution s’élargit en raison d’impulsions aléatoires :
On utilise en exemple cet article :
Solving Fokker-Planck equation using deep learning
avec la comparaison entre analyse et prédiction via ces réseaux neuronaux qui se conjuguent ici :
Le Scale Set d’instances est simplement supprimé ici avec cette ligne :
En conclusion, les méthodes d’apprentissage automatique se sont récemment révélées prometteuses pour la résolution des équations aux dérivées partielles (EDP). Le tout avec une consommation modérée avec ces instances dans Azure :
Avec Julia, il est possible d’aller plus loin notamment en exploitation la capacité des GPU notamment AMD Radeon Instinct puisqu’on a utilisé ici des instances de la série NVv4 d’Azure via notamment AMDGPU.jl (Programmation AMD GPU (ROCm) en Julia).
- GitHub - JuliaGPU/AMDGPU.jl: AMD GPU (ROCm) programming in Julia
- New AMD ROCm™ Information Portal - ROCm v4.5 and Above - ROCm 4.5.0 documentation
En profitant notamment du partage de GPU dans Kubernetes :
- GitHub - RadeonOpenCompute/k8s-device-plugin: Kubernetes (k8s) device plugin to enable registration of AMD GPU to a container cluster
- Kubernetes GPU
À suivre !

















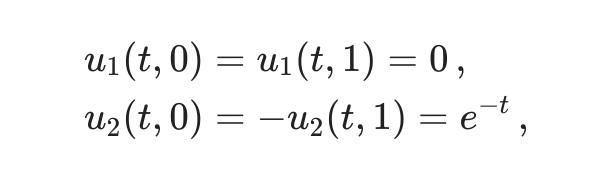











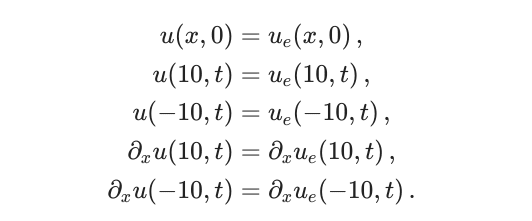







Top comments (0)